A biológiai tudomány doktora, egyetemi tanár
ELTE Növényrendszertani és Ökológiai Tanszék
Miért négybetűs a genetikai ábécé?
A biológusok – s velük az egész tudományos világ – méltán ünneplik a
DNS szerkezetének James Watson és Francis Crick által 1953-ban javasolt
szerkezeti modelljét. Hajlamosak vagyunk az efféle alapvető dolgokat olyannak
elfogadni, ahogy vannak. A Watson-Crick-modell eleganciája szinte sugallja,
hogy az öröklődés anyagi alapja csak így nézhet ki, és nem máshogy. Ezzel
azonban nyilvánvalónak hiszünk valamit, ami korántsem az. Gondoljunk csak
a populációgenetika és a biostatisztika egyik legnagyobb alakjának, Sir
Ronald Fishernek mára klasszikussá vált, 1930-ban megjelent könyvének (A
természetes szelekció genetikai elmélete) előszavában feltett kérdésére:
„Nincsen olyan, a szexuális szaporodás iránt érdeklődő gyakorló biológus,
aki hajlandó lenne rá, hogy kidolgozza három vagy több nemnek az ilyennel
rendelkező szervezetekre vonatkozó részletes következményeit, noha mi mást
kellene tennie ahhoz, hogy megértse, hogy valójában a nemek száma miért
mindig kettő?”. Ezt az útmutatást követve mi is feltehetjük a kérdést:
miért éppen négyféle építőkő szerepel a genetikai anyag kialakításában?
Látni fogjuk, hogy a kérdés és a válasz mára megszűnt merőben spekulatívnak
lenni.
A genetikai ábécé és a négy betű
Mind a DNS-ben, mind az RNS-ben négyféle nukleotid bázist találunk: az előbbiben adenint (A), guanint (G), citozint és timint (T), az utóbbiban timin helyett uracilt (U). Watson és Crick korszakos jelentőségű felismerése szerint ezek kiegészítő párképzéssel tudnak egymáshoz kapcsolódni: az A a T-hez (RNS-ben az U-val) kettő, a G a C-hez három hidrogénhíddal kötődik. Miként már az 1953-as cikkben rámutattak, e szerkezeti sajátosság megalapozza a genetikai anyag másolhatóságát is. Valóban, a DNS-replikáció során a résztvevő polimeráz enzimek az új szálakat a régiek mintáján, a kiegészítő szerkezetet tiszteletben tartva szintetizálják. A genetikai információ az evolúcióban sikeres organizmusok kincse. Nem meglepő, hogy manapság a másolás pontossága igen nagy, a hibahányad 10-10/nukelotid/replikáció körül van. Nagyon régen, több mint hárommilliárd évvel ezelőtt, az élet hajnalán ez nyilván nem lehetett így: az első polimeráz/replikáz enzimek nyilván ennél szerényebb eredményekre voltak csak képesek. A mai másoló enzimek több milliárd éves evolúció termékei. Ebből az is következik, hogy maga a mutációs ráta is evolúciós következmény.
A genetikai ábécé mérete konkrétan felvetodik
Steven Benner és munkatársai rámutattak (Piccirilli et al., 1990), hogy alternatív bázispárok nemcsak elvileg, hanem gyakorlatban is lehetségesek. Mindjárt szintetizáltak is egyet, s kimutatták, hogy a kétféle új nukleotid beépíthető a természetes nukleinsavakba, sőt bizonyos másoló enzimek sikeresen „munkába is veszik” ezeket a replikáció során. Figyeljük, meg, hogy a létező nukleotid bázisok alapvető szerkezete három hidrogénhíd kialakítására ad lehetőséget. Mivel minden hidrogénhídhoz kell egy akceptor és egy donor, és minden bázis lehet kicsi (pirimidin) vagy nagy (purin), ezért elvben 23 nagy és kicsi bázist lehet elképzelni, melyek nyolcféle kiegészítő párba állíthatók (1. ábra).
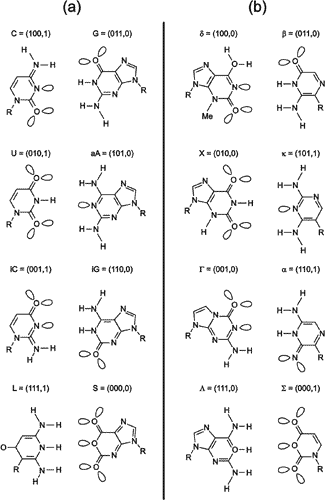
1. ábra. Az elvben lehetséges maximális genetikai ábécé (Mac Dónaill, 2003 nyomán). A természetes genetikai ábécé betűi (az adenin helyett amino-adeninnel) a bal felső sarokban találhatók
Sietve megjegyezzük, hogy kémiai okokból ezek nem mindegyike lehetséges: például nem stabilis egy olyan szerkezet, ahol egyazon gyűrűn három hidrogénhíd akceptor van egymás mellett. Van olyan bázispár, amely szerkezeti okból nagyon sok mutációt indukál. Mindazonáltal e korlátok messze nem szűkítik le a kémiailag is lehetséges szerkezetek körét a természetes genetikai ábécére.
A Nature hasábjain a kommentárt író Leslie Orgel (1990) fel is tette a kérdést: „Miért négybetűs a genetikai ábécé?” Elvben kétféle válasz lehetséges: a biológiai evolúció vagy egyáltalán nem fedezte fel az alternatív bázisokat, vagy valamilyen okból „úgy döntött”, hogy négy betű éppen elég. Újabban egyre több elméleti és kísérleti eredmény mutat abba az irányba, hogy az utóbbi válasz helyes lehet. E cikkben megkísérlem bemutatni a lehetséges válaszok logikáját.
Vissza az RNS-világba
1991-ben és 1992-ben felvetettük annak lehetőségét, hogy a választ az RNS-világban kell keresni. Az RNS-világ egy, az élet korai evolúciójáról szóló hipotézis, mely egyre több alátámasztást nyer. Eszerint volt egy kor, melyben az RNS nemcsak a genetikai állomány, hanem az enzimek szerepét is betöltötte (például Gilbert, 1986). A mai enzimek többsége fehérje. A fehérjék a genetikai információ alapján, húszféle építőkőből (aminosavból) épülnek fel: az aminosavakból kialakuló hosszú lánc bonyolult háromdimenziós térszerkezetet vesz föl. E térszerkezet révén képesek felismerni azokat az anyagokat (a szubsztrátokat), melyek átalakulását gyorsítják. Csakhogy a genetikailag kódolt fehérjeszintézis megjelenése nagyon kemény dió: úgy tűnhet (sokáig úgy is tűnt), hogy nincs genetikai kód fehérjék nélkül és nincs fehérje genetikai kód nélkül. Ez egy klasszikus „tyúk-tojás” probléma, melynek feloldása csakis úgy lehetséges, hogy evolúciósan megvalósítható kis lépésekre bontjuk fel a nagy feladatot, majd megmutatjuk, hogy a kis lépésekre milyen kézenfekvő megoldás van.
Noha sokan (köztük elsőként Carl Woese, Crick és Orgel) spekuláltak arról, hogy esetleg az RNS-nek is lehetnek enzimes sajátságai, erre kísérleti bizonyítékot csak a nyolcvanas évek elején sikerült találni. Crick annak idején a szállító RNS-re (tRNS: a fehérjeszintézis egyik kulcsszereplője, 2. ábra) mondta, hogy „úgy néz ki, mint egy olyan molekula, ami enzimmé akar válni”; a ma ismert enzimhatású RNS-ek (ribozimek) gyakran ennél egyszerűbb szerkezetűek.
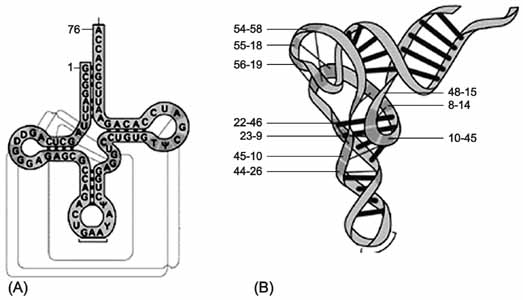
2. ábra. A tRNS másodlagos (a) és harmadlagos (b) szerkezete. A másodlagos szerkezetet Watson-Crick típusú bázispárok alakítják ki, a harmadlagos szerkezetet más jellegű, „extra” hidrogénhidak rögzítik
A legtöbb, mai organizmusból izolálható ribozim más nukleinsavak átalakítását végzi. Csakhogy ettől még nem lesz teljes az RNS-világ! A nukleinsavak építőköveinek szintézisét biztosító anyagcsere lépéseiről is gondoskodni kellene (Benner et al., 1989). Ma ilyen természetes ribozimet nemigen ismerünk: ha voltak is valaha, a fehérjék helyettesítették őket. De lehettek-e egyáltalán? Sokan kételkedtek ebben. Korábban felvetettük annak lehetőségét, hogy kémcsőben folytatott mesterséges szelekció révén előre meghatározott aktivitású ribozimeket gyárthatnánk (Szathmáry, 1990). Ez a mag meglepően erős szárba szökkent: mára a kismolekulákhoz sikeresen kötődő és azokat átalakítani tudó ribozimekből több tucatot ismerünk. Úgy tekinthetünk ezekre, mint a hajdani RNS-világ résztvevőivel analóg szerkezetekre. Még nem tudjuk azonban, mi köze mindennek a genetikai ábécéhez?
Egy „ribocita” rátermettsége
A rátermettség az evolúcióbiológia egyik központi fogalma: az utódok várható számát jelenti az organizmus élettartama során. A ribocita egy fehérjék helyett ribozimekkel működő (hipotetikus) sejt. Feltettük a kérdést: miként alakul egy ribocita rátermettsége a genetikai ábécé méretének függvényében (Szathmáry, 1991, 1992)?
A válaszhoz vissza kellett nyúlni Manfred Eigen 1971-es dolgozatához,
melyben a rátermettséget (W) két komponensre osztotta fel: a szaporodási
sebességre (A) és a genetikai üzenet másoldódásának pontosságára
(Q), vagyis W = AQ. Feltételezésünk szerint mindkettő függ
a genetikai ábécé N méretétől, vagyis W(N) = A(N)Q(N). Nem
marad más hátra, mint tisztázni, hogy is néz ki A(N) és Q(N)!
A szaporodási sebesség nő az ábécé méretével
Mint láttuk, az enzimek bonyolult szerkezetű makromolekulák. A szerkezet részletei egymáshoz illetve a szubsztráthoz az építokövek szerkezeti gazdagságát kihasználva finoman illeszkednek. Minél finomabb az illeszkedés, annál jobb a funkció. Igen ám, de kevesebb fajta építőkő átlagosan kisebb hatékonyságot jelent. Nyilván ez volt a fő oka annak, amiért a húszféle aminosavból felépülő fehérjék átvették a négyféle nukleotidból felépülő ribozimek helyét. Most azonban gondolatban még az RNS-világban vagyunk. A mondottakból következik, hogy A növekvő függvénye N-nek. De pontosan hogyan növekszik?
Ha a szubsztrátok kötésének átlagos erossége lineárisan növekszik, akkor lineárisan csökken az aktiválási energia, és ez által gyorsul a reakció. Az aktiválási energia csökkentése és a reakciósebesség között viszont a fizikai kémia szerint exponenciális összefüggés van. Tegyük fel, hogy sokféle ribozimre átlagosan teljesül a következő feltétel: minél többféle betűvel dolgozunk, annál finomabb szerkezeteket építhetünk, de érvényesül a csökkenő hozadék törvénye is, hiszen a bázisok egyre inkább hasonlítani fognak a meglévőkre (vö. 1. ábra). Feltételezzük továbbá, hogy a ribozimek átlagos katalitikus aktivitásnak növekedése lineárisan csökkenti a ribocita osztódási idejét. Az átlagos aktiválási energia csökkenés tehát N-ben a lineáris alatt marad (telítési görbe szerint), miáltal A az N-nel a mondottak miatt a lineárisnál gyorsabban, az exponenciálisnál viszont lassabban növekszik.
Van-e erre valamilyen kísérleti bizonyíték? Szerencsére akad. A már említett in vitro szelekció útján Gerald Joyce laboratóriumában sikerült olyan ligáz funkciójú (két másik nukleinsav darabot összekapcsoló) ribozimeket előállítani, melyek közül az egyik háromféle (Rogers – Joyce, 2001), a másik csupán kétféle nukleotidból (Reader – Joyce, 2002) áll. Az első érdekes következtetés az, hogy még a kétféle nukleotidból (A és U) álló ribozim is mintegy 36 000-szeresére növeli a reakció sebességét. A másik konklúzió pedig az, hogy a katalitikus hatékonyság az exponenciálisnál lassabban növekszik. Valójában a növekedés még a lineárisnál is gyengébb, de ennek oka valószínűleg az, hogy ebben az esetben az ábécé méretének csupán indirekt hatása volt a katalízisre amennyiben nem az aktív centrum jóságát, hanem a ribozim „állványzatának" milyenségét befolyásolta csak. Kíváncsian várjuk az újabb, a szisztematikus tesztelést célzó eredményeket.
A másolódási hűség csökken az ábécé méretével
A másolódási hűség csökkenésének intuitíve világos alapja megint csak az, hogy minél többféle betű van (1. ábra), annál könnyebb őket összetéveszteni. Ennek igaznak kell lennie – általában – a polimeráz/replikáz enzimekre is, és ennek mennyiségi kifejezést lehet adni. Ismét csak Eigen (1971) nyomán feltételezzük, hogy egy n hosszúságú genetikai üzenet másolódási hűsége a betűnkénti másolási pontosság (q) függvénye, vagyis Q = qn, ahol feltételezzük, hogy valamely nukleotid másolódási pontossága független a szomszédokétól. Számoljunk mintegy 104 hosszúságú genommal. Milyen alakú lesz a q(N) függvény?
A mutációkról szerzett ismeretek szerint érvényes ama összefüggés, miszerint a q a megfelelő és a rossz (mutáns) párok kötődésének mértékétől függ. Ez pedig a rossz és a jó formák, mint megfordítható reakciók viszonylagos egyensúlyi állandójának a függvénye. Ez utóbbi viszont exponenciálisan függ a kötési energiák különbségétől, ami a hiperkocka betöltése miatt egyre kisebb differenciákat jelent a jók javára. Summa summarum, azt kapjuk, hogy a Qaz exponenciálisnál gyorsabban csökken a genetikai ábécé méretével.
Ez a jóslat elvben tesztelheto; a baj csak az, hogy gyakorlatban a létezo enzimek (polimerázok és replikázok) nem működnek elég általánosan ahhoz, hogy a maximális ábécé-másolási hűségét vizsgálni tudjuk. Arról nem is beszélve, hogy tulajdonképpen replikáz funkciójú ribozimre lenne szükségünk; ilyesmi azonban – kellően hatékony formában – még egyáltalán nem áll rendelkezésünkre. A q(N) függvény alakjának tesztelése még várat magára.
Ennek ellenére az elméleti üzenet világos. Minthogy W = AQ, ezért egy ribocita rátermettsége valamilyen N értéknél maximumot kell hogy mutasson! Erre utaltunk akkor, amikor a genetikai ábécé méretét az RNS-világ befagyott örökségének tekintettük (Szathmáry, 1991).
Váratlan hozzájárulás a kódolás elméletéből
Úgy fél évvel ezelőtt Dónall Mac Dónaill (2002, 2003) elméleti kémikus friss szemmel tekintett az ábécé méretének kérdésére. Felismerte, hogy a lehetséges nukleotidok kódolhatók – ez a kódolás nem tévesztendő össze a genetikai kóddal, ami a bázisok hármasa és az aminosavak megfeleltetése a fehérjeszintézis folyamatában – zérusok és egyesek egy vektorával, ahol a hidrogén-donor csoportok mindig 1, a hidrogén-akceptor csoportok mindig 0 értéket kapnak, a kicsi nukleotid kódja 1, a nagyé pedig 0 a vektor negyedik helyén (3. ábra).
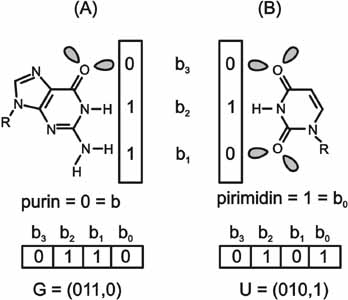
3. ábra. A citozin és a guanin kódolása Mac Dónaill (2002) szerint
Ezek után definiálható az nukleotidok ún. paritása, vagyis hogy a kódjukban hány darab egyes van összesen. Nézzük meg a jelenlegi genetikai ábécét (itt az egyszerűség kedvéért az adenint az amino-adeninnel [aA] helyettesítjük)! A kódok a következők: C=(1001), G=(0110), U =0101), aA=(1010). A kiegészítő párképzés szabályai szerint egy tökéletes bázispár két tagjának a kódoló vektora is kiegészítő: egyessel szemben zérus, zérussal szemben egyes áll. Ez még nem különösen mély felismerés. Az már jóval érdekesebb, hogy a paritások alapján definiálhatunk tisztán páros és tisztán páratlan paritással rendelkező ábécéket (4. ábra).
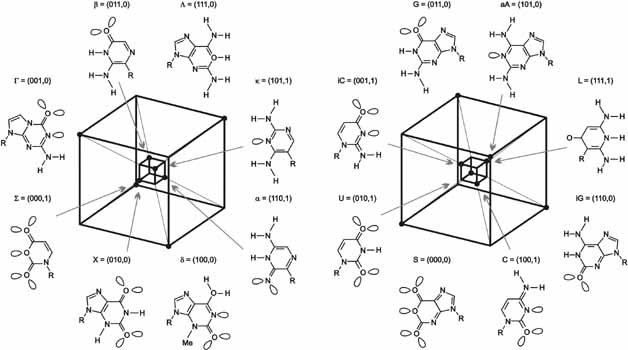
4. ábra . Mac Dónaill (2002) bevezetette a tiszta páratlan (a) és páros (b) paritású genetikai ábécék halmazát. Mindkettő egy hiperkockán helyezhető el, a nagy bázisok kívül, a kicsik belül helyezkednek el. A természetes genetikai ábécé a jobb oldali, tiszta páros paritású teljes ábécé egy részhalmaza
Figyeljük meg, hogy a természetes genetikai ábécé tiszta, páros paritású képződmény.
Mire is jók a tiszta paritású ábécék? Mac Dónaill felismerése szerint ezek minimalizálják a mutációs rátát, vagyis maximalizálják a Q-t! Ez azért van így, mert a tiszta paritású ábécékben a jó és a rossz beülő nukleotid legalább 2 kódolt bitben különbözik egymástól. Ez érvényes a C és az U, illetve az A és a G viszonyára (a leggyakoribb mutációk azonos méretű nukleotidot ültetnek be rosszként; ezeket tranzícióknak nevezzük). Éppen ilyen ábécét javasolna a hibajavító kódolási eljárások szakértője is! Két bitben egyszerre tévedni jóval valószínűtlenebb, mint egyben. A kevert paritású ábécék nagyon rosszak azért, mert bennük a jó és a rossz között egyetlen bitnyi differencia is megengedett. Ez annyit jelent, hogy csupán egyetlen nemkötő vagy taszító hidrogénhíd pozícióban van eltérés. Ha most visszatérünk az energetikára, kiszámolható, hogy ebben az esetben a megfelelő és rossz párok közötti kötési differencia olyan kicsiny, hogy a másolási hűság is reménytelenül kicsi lesz.
Mi szűkíthette tovább a lehetőségek körét? Már említettem, hogy
például a 000 mintázat (három akceptor) kémiailag nem szintetizálható.
Ezen felül az izocitozin-izoguanin (iC-iG) pár olyan gyakran alakul át
egy másik formába, hogy korábban a mesterséges mutagenezisben használták.
A korai evolúcióban, ahol nem a mutációk, hanem a pontos másolatok számának
gyarapítása volt a fő cél, egy ilyen pár nem „rúghatott labdába”. Ha viszont
az iC-iG pár ki van zárva, akkor – a részletes energetikai számolás szerint
– az amino-adenin nem nyújt számottevő előnyt az adeninnel szemben! Úgy
tűnik tehát, hogy az evolúciónak, ha a Q-t maximalizálta, akkor tiszta
paritású ábécé mellett kellett kikötnie. Ha ezen belül páros paritású ábécé
mellett „döntött”, akkor a jelenlegi természetes ábécé az egyetlen megoldás.
Az, hogy miért nem egy páratlan paritású ábécé alakult ki, ma még nem tisztázott
kérdés. Itt valóban szerepet játszhatott a bázisok korai hozzáférhetősége.
Az adenin mint a hidrogén-cianid ötszörös „polimerje”, például igen könnyen
keletkezik ammónia jelenlétében e mérgező vegyületből. A kezdeti szimmetriát
tehát egy ilyen egyszerű hatás is megsérthette.
Egy másik szempont: az evolúcióképesség
Az evolúcióképesség (evolvabilitás) a genetikai rendszerek olyan sajátossága,
amely hosszú távon befolyásolja ezek evolúciós sikerét a változó környezethez
való idomulás elősegítésén keresztül. Sokan úgy gondolják, hogy a szexuális
szaporodás általában fokozza a populációk evolúcióképességét.
Nos, több vizsgálat azt jelzi, hogy a különböző genetikai ábécékkel
épített replikátorok evolúcióképessége eltérő. Ezt a kutatási programot
a bécsi elméleti biológiai iskola indította el (Schuster, 1993; Hofacker
et al., 1994), s legújabban új-zélandi kutatók fejlesztették tovább (Gardner
et al., 2003).
Mielőtt néhány részletet ismertetnék, érdemes kitérőt tenni az RNS lehetséges szerkezeteire. Miként a tRNS-eknek is, általában az RNS-eknek is van két- és háromdimenziós szerkezete. A kétdimenziós szerkezet a láncon belüli párképzés eredménye. Az esetleges funkciót a háromdimenziós (harmadlagos) szerkezet határozza meg, ezt azonban – a fehérjéhez hasonlóan – még nem tudjuk kiszámítani pusztán az építőkövek sorrendjéből. Másodlagos szerkezet számítására azonban igen hatékony algoritmussal rendelkezünk, mely a legstabilisabb struktúrá(ka)t keresi meg, vagyis amelyben a legtöbb erős bázispár képződik (Zuker, 2000). Az evolvabilitással kapcsolatos vizsgálatok sok szekvencia másodlagos szerkezetének összehasonlításán és statisztikai elemzésén alapulnak.
Lássuk először a szerkezetek egyfajta statikus összehasonlítását! Könnyen belátható, hogy a kevés, például kétféle nukleotidból felépülő molekulák könnyen billennek át másfajta másodlagos szerkezetbe, mert nagyon sok a párosodási lehetőség (ezt a korábban említett, csak kétféle nukleotidból álló ribozim esetén is látszott). Minél többféle betű van, annál jobban stabilizálhatók az egyedi struktúrák, vagyis annál több mutáció kell ahhoz, hogy egy molekula másodlagos szerkezet az egyikből egy másik alakba átbillenjen. Ez nyilván kedvez a már elért szerkezet stabilitásának, de – érezhető módon – lassítja az evolúciót. Ezt a sejtést Gardner és munkatársai (2003) számítógépes evolúciós kísérletek segítségével ellenőrizték. Egy tRNS-szerű struktúra kialakulásának sebességét vizsgálták véletlenszerű szekvenciák szimulált mutációja és szelekciója útján. Azt találták, hogy nem túl pontos q esetén a négybetűs ábécét használó molekulák érték el a kitűzött célt a leggyorsabban.
A jövő
A jövő nyilván a lehetőségek rendszeres és minél általánosabb feltételek közötti vizsgálatát hozhatja. Mint már említettem, szükség lesz kiterjesztett ábécéből épülő molekulákat másolni tudó replikáz enzimekre. A genetikai ábécének a metabolizmus sebességére tett hatása sokféle, különböző aktivitású ribozim összehasonlításán mérhető csak le megbízhatóan.
E vizsgálatok választ adhatnak ama kérdésre is, hogy vajon a földitől függetlenül kifejlett élővilágok genetikai anyaga milyennek várható. Azt sokan gondolják, hogy egy fejlett öröklődési rendszer mindig digitális (vagyis betűk sorrendjén alapszik), de hogy hányféle betűből áll, azt kevesen kérdezték és próbáltak meg rá válaszolni. Ha például a Jupiter Európa nevű holdján létezik egy, a földitől független élővilág, akkor ennek jövőbeni vizsgálata konkrét választ adhat e kérdésre. Addig is azonban sokat megtudhatunk az elméleti és kísérleti módszerek fejlesztésével.
Van egy párja a genetikai ábécé kérdéskörének, s ez a katalitikus
ábécé méretének a kérdése. Az RNS-világ korában e kettő egybeesett, ma
azonban az utóbbi a fehérjéket felépítő aminosavfélék számára vonatkozik.
Nem kizárt, hogy az itt bemutatottakhoz hasonló gondolatok – alkalmas kiegészítésekkel
– erre a kérdésre is választ adnak majd.
Irodalom
Benner, Steven A. – Ellington, Andrew D. – Tauer, Andreas (1989): Modern Metabolism as a Palimpsest of the RNA World. Proceedings of the National Academy of Sciences of the USA. 86, 7054–7058.
Crick, Francis H. C. (1968): The Origin of the Genetic Code. Journal of Molecular Biology. 38, 367–379.
Eigen, Manfred (1971): Self-organization of Matter and the Evolution of Biological Macromolecules. Naturwissenschaften. 58, 465–523.
Fisher, Ronald A. (1930): The Genetical Theory of Natural Selection. Clarendon, Oxford
Gardner, Paul P. – Holland, B. B. – Moulton, V. – Hendy, M. – Penny, D. (2003): Optimal Alphabets in an RNA World. Proceedings of the Royal Society of London Series B. in press.
Gilbert, Walter (1986): The RNA World. Nature. 319, 818.
Hofacker, Ivo L. – Fontana, W. – Bonhoeffer, S. – Stadler, P. F. (1994): Fast Folding and Comparison of RNA Secondary Structures. Monatshefte für Chemie, 125, 167–188.
Mac Dónaill, Dónall (2002): A Parity Code Interpretation of Nucleotide Alphabet Composition. Chemical Communications, 18, 2062–2063.
Mac Dónaill, Dónall (2003): An Error-coding Perspective of Nucleotide Alphabet Composition. Origins of Life and Evolution of the Biosphere. in press
Maynard Smith, John – Szathmáry Eörs (1995): The Major Transitions in Evolution. Freeman & Co., Oxford.
Orgel, Leslie E. (1968): Evolution of the Genetic Apparatus. Journal of Molecular Biology. 38. 381–393.
Orgel, Leslie E. (1990): Adding to the Genetic Alphabet. Nature. 343, 18–20.
Piccirilli, Joseph A. – Krauch, T. – Moroney, S. – Benner, S. (1990): Enzymatic Incorporation of a New Base Pair into DNA and RNA Extends the Genetic Alphabet. Nature, 343, 33–37.
Reader, John S. & Joyce, Gerald F. (2002): A Ribozyme Composed of Only Two Different Nucleotides. Nature, 420, 841–844.
Rogers, Jeff & Joyce, Gerald F. (2001): The Effect of Cytidine on the structure and Function of an RNA Ligase Ribozyme. RNA. 7, 395–404.
Schuster, Peter (1993): RNA Based Evolutionary Optimization. Origins of Life and Evolution of the Biosphere. 23, 373–391.
Szathmáry Eörs (1990): Towards the Evolution of Ribozymes. Nature. 344, 115.
Szathmáry E. (1991): Four Letters in the Genetic Alphabet: A Frozen Evolutionary Optimum? Proceedings of the Royal Society of LondonSeries B. 245, 91–99.
Szathmáry Eörs (1992): What Determines the Size of the Genetic Alphabet? Proceedings of the National Academy of Sciences of the USA. 89, 2614–2618.
Woese, Carl R. (1967): The Genetic Code. Harper & Row, New York
Zuker, Michael (2000): Calculating Nucleic Acid Secondary Structure. Current Opinion in Structural Biology, 10 (3), 303–310.
<